User:Defuneste/Les arbres isolés dans OSM
Les arbres isolés dans OSM
Dans le cadre de l'ANR ReVeRIES je suis amené à travailler sur les arbres isolés et plus largement sur les arbres dans OSM. Après discussion avec le groupe lyonnais il a été décidé de faire une page pour garder les discussions.
Les graphiques et statistiques sont issus d'un export d'octobre 2018 de la zone française à partir de geofabrik, puis de début novembre 2018 via Data Gouv. PostgresSQL (10) et R (3.4.4) sont utilisés, tous les deux en local. Les scripts des stats sont sur le github du projet. L'analyse est en cours, cette page évoluera donc le long des discussions et des apports. Les différentes cartes et graphiques sont faits pour de l'analyse et exploration de données donc du "laid et rapide". Ce travail est fait en parallèle avec l’étude de faisabilité des imports d'arbres dans OSM et il peut être utile de jeter à mes autres pages.
Une rapide introduction des arbres dans OSM
Les arbres isolés sont référencés par des nœuds associés à un attribut natural=tree. La page wiki française est un peu en retard mais la page anglaise explique bien les principaux attributs et les valeurs qu'ils peuvent prendre. On verra ultérieurement que ces consignes ne sont pas toujours suivies et il a été mentionné que les différentes plateformes de cartographie ne permettaient pas toujours le bon renseignement selon les règles communes.
Avec une sélection sur natural=tree dans la table des nœuds (planet_osm_point) on obtient 8880626 arbres. Ces arbres sont décrits par 29 attributs (en plus de 'tags' et 'natural') complétés par 162 champs dans tags (la colonne de typmod hstore dans postgres). Cela fait donc 191 attributs, rien que pour les arbres isolés.
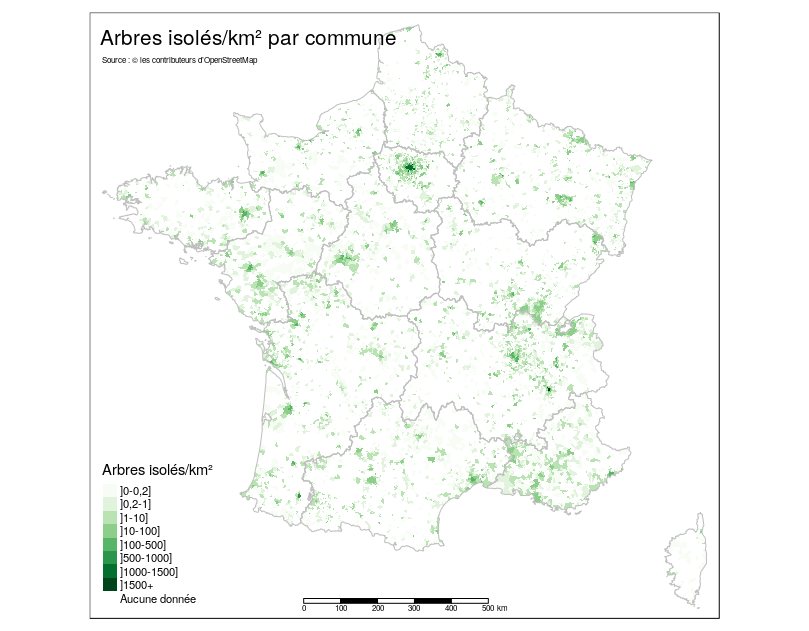
La carte suivante reprend le nombre de ces arbres par commune et par km²
La clef genus
Cette clef est utilise pour indiquer le nom de genre en latin et la première lettre doit être capitalisée. Il y a 81230 renseignements dans "genus" pour 265 valeurs différentes soit 9,2% des arbres isolés.
species.shp <- st_read(con, query =
"SELECT way, tags -> 'species' AS species, tags -> 'genus' AS genus
FROM planet_osm_point
WHERE planet_osm_point.natural = 'tree';")
species.dat <- st_set_geometry(species.shp, value = NULL) # drop de geometry
#un tableau pour explorer
sort(table(species.dat$genus), decreasing = T))
# genres commencant par une minuscule
unique(species.dat$genus)[grep(pattern = "^[[:lower:]]",
unique(species.dat$genus))]
# genres contenant un espace
unique(species.dat$genus)[grep(pattern = "\\s", unique(species.dat$genus))]
# genres contenant un ;
unique(species.dat$genus)[grep(pattern = "\\;", unique(species.dat$genus))]
Un rapide coup d’œil au tableau des décomptes de nombres d'arbres par genre indique des erreurs :
- les noms de genre mais non capitalisés ("abies"),
- les noms de genre avec un accent ("oléa"),
- les noms en français ("chênes"),
- les erreurs de codage/import ("Acer;Aesculus" ou "genus:fr=Tilleul"),
- les noms de taxons/d'espèce ("Tilia platyphyllos scop", "Picea abies"),
- des choses curieuses ("genus", "inc", "palm")
C'est assez difficile d'automatiser les vérifications tant que l'on a pas une liste exhaustive des genres des requêtes et fonctions ont été écrites plus bas (partie "code").
Petit bilan temporaire sur Genus :
tableau_selection("\\;|è|é|ê|^[[:lower:]]|\\s")
Sur les 80170 renseignements dans "genus" pour 265 valeurs on trouve au moins 1492 lignes avec des erreurs (moins de 2%) pour 115 valeurs distinctes. Ce nombre d'erreurs peut egalement être fortement diminué automatiquement en comparant à un "genus" capitalisé présent ou en comparant à un "species" présent (dans le cas de plusieurs mots).
Le nombre d'erreurs influe cependant fortement sur le nombre valeurs correctes, ce nombre doit approcher les 150.
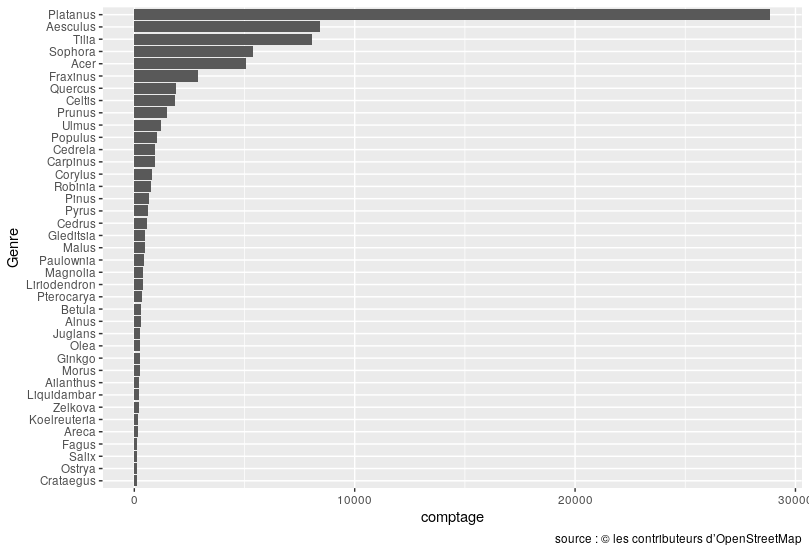
Le graphique ci dessous présente les principaux genres (en dehors des valeurs manquantes et des genres de moins de 100 arbres). Les platanes sont les plus renseignés, puis les marronniers et tilleuls. Les sophora et érables viennent ensuite les frênes, les chênes, les micocouliers et les prunus.
Il y a 14786 arbres isolés avec un genre ayant également un champ "species" rempli.
Species
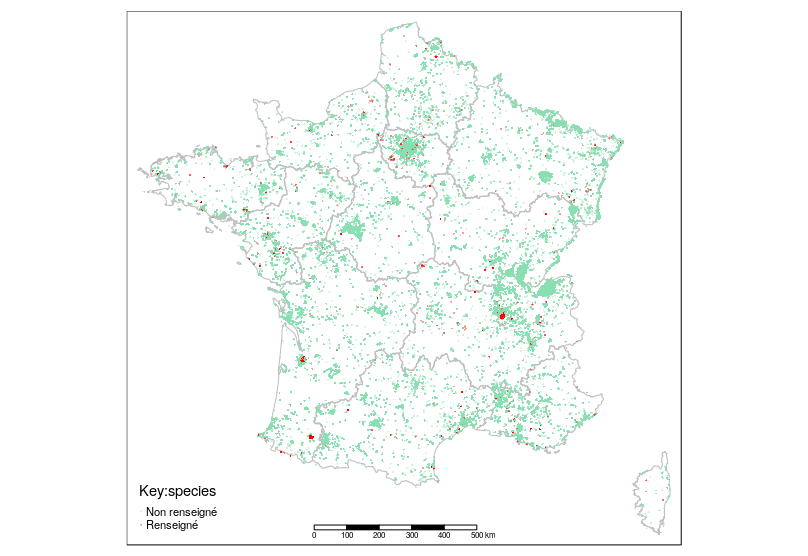
L'attribut "species" est le plus renseigné avec 99032 valeurs non nulles (près de 12% du nombre d'arbre). Il y a 1814 valeurs différentes. La répartition des arbres isolés se rapproche des centres urbains (et peut-être des communautés de contributeurs ?) à la différence du renseignement du champs "species" qui ne semble pas suivre tout à fait cette logique.
Le wiki indique que "species" doit correspondre au nom latin (binomial). On trouve plusieurs écarts à cette règle :
- Les cas d'une information plus précise : le cultivar ou la variété de mentionné (avec plusieurs syntaxes possibles)
- Les cas de nomenclatures différentes : première lettre du descripteur
- Les erreurs :
- des plantes non ligneuses : "iris versicolor" par exemple ;
- uniquement le nom du genre : "platanus" / "Platanus" ;
- des noms vernaculaires, dans des langues variées : "Olivette blanche" ;
- des choses étranges : "OEIL" ;
- etc.
Un exemple sur les "platanes" :
Il y a 32 descripteurs (cf. tableau plus bas) couvrant 15350 "platanes" dans l'attribut "species". Certaines différences peuvent êtres dues à des caractères spéciaux n’apparaissant pas.
La grande majorité est représentée par "Platanus acerifolia" et "Platanus x hispanica" qui sont synonymes.
Les classifications botaniques évoluent et, afin de savoir où en sont les botanistes, ces sites ont été utilisés : TRNS et Tela Botanica.
On trouve plusieurs types d'erreurs :
- Les noms de genre : Le nom de genre devrait être mis dans l'attribut Key:genus. Selon le wiki OSM pour ce dernier, il doit être en latin et la première lettre capitalisée. On retrouve dans cette catégorie : platanus, Platanus, platane, platanes, Platanus sp ou encore Platanus n. sp.
- Les fautes de frappe de plusieurs types :
- L'absence de capitalisation du genre, la capitalisation du nom d'espèce et celle du croisement (moins tranché)
- La coquille : Platanus aceriofolia
- Le non respect de la consigne du wiki : nom vernaculaire plutôt que nom latin
Il y a ensuite le cas de synonymes et des différences de nomenclature en botanique. Il faut rappeler que le nom latin peut avoir évolué : un arbre a pu être décrit dans un pays et dans un autre avec des différences qui ne sont plus reconnues aujourd'hui. Le nom latin peut être suivi d'une lettre majuscule suivie d'un point (L. pour Linné) qui correspond au premier descripteur de l'espèce reconnue (Platanus orientalis L.). Cet usage, avec parfois la date de la description associée, est plus de rigueur dans des travaux de botanique où il peut y avoir besoin de spécifier et où les lecteurs peuvent apprécier la différence.
Le cas des platanes comporte des cultivars (ex : Platanus orientalis 'Digitata' : le nom du cultivar doit être entre '' et la première lettre capitalisée) que l'homme a produit mais (à ma connaissance) pas de variété produite sans intervention humaine (Phillyrea angustifolia media par ex). Dans OSM ce niveau de précision est réservé à taxon. Le taxon peut à la fois correspondre à des catégories fines (cultivars, variétés) ou à une catégorie plus englobante comme rosacées (qui est une famille).
Platanus acerifolia et Platanus x hispanica sont des synonymes. C'est un croisement entre Platanus orientalis et Platanus occidentalis. Le × indique le croisement, c'est le symbole multiplié (× en HTML) qui peut parfois être noté x en minuscule et pas italique (source).
"Platanus platanor" doit correspondre à un certificat d'obtention végétale qui correspond à un hybride cloné 'Vallis clausa'. Ce n'est pas clair s'il doit rentrer dans Plantanus x hispanica en espèce puis Plantanus x hispanica 'Vallis clausa'.
| Descripteurs | Descompte | Type d'erreur |
|---|---|---|
| Platanus acerifolia | 8737 | synonyme |
| Platanus x hispanica | 5456 | ok |
| Platanus orientalis | 243 | ok mais rare |
| Platanus x acerifolia | 231 | synonyme |
| platanus | 193 | nom de genre |
| Platanus platanor | 79 | taxon |
| Platane | 75 | nom de genre |
| Platanus hispanica | 71 | Platanus x hispanica |
| Platanus ×hispanica | 57 | coquille |
| Platanes | 42 | nom de genre |
| Platanus | 40 | nom de genre |
| platane | 32 | nom de genre |
| Platanus ×acerifolia | 25 | coquille |
| Platanus xacerifolia | 13 | coquille |
| Platanus orientalis Digitata | 7 | taxon |
| Platanus x acerfoilia | 7 | synonyme |
| Platanus X acerifolia | 7 | X ou x |
| Platanus X hispanica | 7 | X ou x |
| Murier Platane | 6 | nom vernaculaire, murier |
| Platanus occidentalis | 6 | ok |
| Platanus orientalis L. | 3 | plus spécifique |
| Platane à Feuille d'Érable | 2 | nom vernaculaire |
| Platanus n. sp. | 2 | nom de genre |
| platane commun | 1 | nom vernaculaire |
| platanoides | 1 | érable ? |
| Platanus × acerifolia | 1 | synonyme |
| Platanus aceriofolia | 1 | coquille |
| Platanus Orientalis | 1 | coquille (maj à l'épithète) |
| Platanus orientalis 'Digitata' | 1 | taxon |
| Platanus sp. | 1 | nom de genre |
| Platanus x Hispanicus | 1 | coquille (hispanica) |
| platanus xacerifolia | 1 | coquille |
Quel niveau de précision doit on souhaiter ? C'est à débattre, mais je ne pense pas que l'on puisse demander aux contributeurs de répondre à des questions qui peuvent faire débats chez les botanistes et qui si besoin peuvent faire l'objet de traitement au niveau de l'analyse (rassembler les synonymes par exemple). Par contre, je pense que c'est pédagogique (du point de vue botanique) de faire la différence entre un genre (Platanus) et une espèce (Platanus × hispanica) et donc d'utiliser leurs clefs respectives.
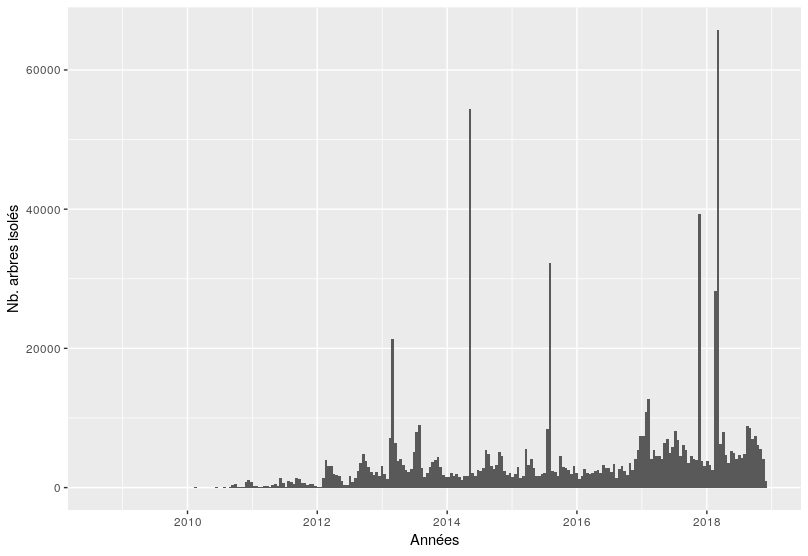
Dynamique des contributions autour des arbres isolés :
Répartition des arbres isolés en France métropolitaine :
En fonction de Rural/Urbain :
Les catégories rural/urbain sont celles utilisées par l'INSEE :
Arbres isolés présents dans des communes rurales : 125510/422721,3 km2 (0.3 arbres isolés/km2)
Arbres isolés présents dans des communes urbaines : 694969/126164,6 km2 (5,51arbres isolés/km2)
vérifier: "Emplacement à planter" dans espèce
Code alourdissant la lecture :
Pour genus :
Erreurs détectées via les accents:
tableau_selection <- function(patron_regex, colonne="genus") {
filtre <- unique(species.dat$genus)[grep(pattern = patron_regex, unique(species.dat[[colonne]]))]
decompte <- species.dat %>%
filter(genus %in% filtre) %>%
group_by(genus) %>%
summarize(comptage = n()) %>%
arrange(desc(comptage))
# impression du tibble
print(decompte)
# impression du nombre de ligne concernées
sum(decompte$comptage)
}
tableau_selection("(\\è|é|ê)")
Une fonction permet d'afficher un tableau et la somme des erreurs pour une expression régulière. Dans ce cas, il y a 278 lignes avec des accents repartis sur 11 genres. Ici pas d'idée pour corriger automatiquement, il faut le faire à la main.
Erreurs détectées via la non capitalisation :
Ici l'objectif est de regarder si l'on peut trouver un moyen de corriger les simples erreurs de capitalisation de lettre. On part de l’idée que la bonne écriture est déjà dans OSM et on va comparer les valeurs de genus non capitalisées avec celles capitalisées. Une fonction permet de le faire l'argument negation indique avec TRUE si on retrouve le mot non capitalisé dans ceux capitalisé et FALSE si on ne le retrouve pas.
tableau_selection_capital <- function(patron_regex, negation = FALSE) {
genus_upper <- str_to_title(unique(species.dat$genus)[grep(pattern = patron_regex, unique(species.dat$genus))])
if(negation == TRUE){
genre <- genus_upper[genus_upper %in% unique(species.dat$genus)]
decompte <- species.dat %>%
filter(genus %in% str_to_lower(genre)) %>%
group_by(genus) %>%
summarize(comptage = n()) %>%
arrange(desc(comptage)) }
else {
genre <- genus_upper[!genus_upper %in% unique(species.dat$genus)]
decompte <- species.dat %>%
filter(genus %in% str_to_lower(genre)) %>%
group_by(genus) %>%
summarize(comptage = n()) %>%
arrange(desc(comptage)) }
print(decompte)
sum(decompte$comptage)
}
tableau_selection_capital("^[[:lower:]]", negation = T)
tableau_selection_capital("^[[:lower:]]", negation = F)
| Genus | nbr |
|---|---|
| marronnier | 26 |
| cupressus sempervirens | 20 |
| mimosa | 9 |
| pin | 8 |
| palm | 6 |
| oak | 5 |
| willow | 4 |
| coniferous | 3 |
| cèdres | 2 |
| coniferus | 2 |
| broadleaved | 1 |
| conifer | 1 |
| erable | 1 |
| inc | 1 |
| marronier | 1 |
| palmae | 1 |
| séquoia géant | 1 |
| syringa | 1 |
Il y 93 erreurs (en comptant cèdres et séquoia géant). On peut proposer des corrections pour certaines ("marronier" = Aesculus, "cupressus sempervirens" = Cupressus par exemple). D'autres sont trop imprécises pour proposer des alternatives ("broadleaved", "conifer"). Ensuite "syringa" (Syringa), est un nom de genre valable mais il est le seul présent donc on ne l'obtient donc pas capitalisé.
Ensuite on vérifie l'inverse : une fois capitalisé retrouve-t-on un bon nom de genre ?
| genus | nbre |
|---|---|
| pinus | 178 |
| prunus | 53 |
| olea | 51 |
| cupressus | 46 |
| platanus | 43 |
| platane | 31 |
| tilia | 30 |
| areca | 29 |
| quercus | 21 |
| celtis | 20 |
| taxus | 18 |
| chêne | 17 |
| morus | 16 |
| ficus | 10 |
| malus | 10 |
| cedrus | 9 |
| fagus | 6 |
| magnolia | 6 |
| olivier | 4 |
| phoenix | 3 |
| sequoia | 2 |
| séquoia | 1 |
| abies | 1 |
| betula | 1 |
| castanea | 1 |
| populus | 1 |
| pyrus | 1 |
| sequoiadendron | 1 |
| tamarix | 1 |
On peut voir 4 erreurs (dont 35 non encore identifiées) avec cette façon de procéder (en gras dans le tableau): il doit exister des "Olivier", "Platane", "Chêne" et "Séquoia" (ces deux dernières apparaissaient déjà) et donc ces noms sont gardés alors qu'il devrait être indiqué en "mauvais_genre".
Si on peut corriger en amont ces genres la méthode permet de sortir pas mal d'erreurs.
Erreurs avec plusieurs mots dans genus :
tableau_selection_essence <- function(patron_regex, negation = FALSE) {
genre <- unique(species.dat$genus)[grep(pattern = patron_regex, unique(species.dat$genus))]
if(negation == TRUE){
genre <- genre[genre %in% unique(species.dat$species)]
decompte <- species.dat %>%
filter(genus %in% genre) %>%
group_by(genus) %>%
summarize(comptage = n()) %>%
arrange(desc(comptage)) }
else {
genre <- genre[!genre %in% unique(species.dat$species)]
decompte <- species.dat %>%
filter(genus %in% genre) %>%
group_by(genus) %>%
summarize(comptage = n()) %>%
arrange(desc(comptage)) }
print(decompte)
sum(decompte$comptage)
}
tableau_selection_essence("\\s", negation = T)
tableau_selection_essence("\\s", negation = F)
On obtient 545 arbres pour 55 valeurs. Si on regarde les noms de genre avec un espace on trouve pas mal de nom ressemblant à des noms d'espèces (genus ayant été remplie en plus ou à la place de species). Est-il possible comme pour les noms de genre non capitalisées de les retrouver dans le champs species ?
La ligne sortant les noms non présent retourne cette liste : "Carpinus betulu", "Olea european", "séquoia géant", "cupressus sempervirens","Robinia pseudacacia" , "Calocedrus Decurrens Flo.", "Sequoiadendron giganteum var. pendulum", "Tilia platyphyllos scop", "Sophora Japonica", "Sorbus domestica L.", "Cedrus Libani A. Rich." (11 valeurs pour 58 lignes)
Elle semble recouvrir 5 cas : les lettres en moins/coquilles ("Carpinus betulu" au lieu de Carpinus betulus, "Olea european"/Olea europaea), le manque de capitalisation ("cupressus sempervirens"), un nom latin "enrichi" ("Sorbus domestica L."), un nom latin correcte mais que l'on ne retrouve pas ("Sophora Japonica") et un nom d'espèce en français ("séquoia géant", déjà observé). Les noms latins "enrichi" auraient leur place sans la clef taxon et une extraction permettrait d'en tirer et garder le nom latin.
La seconde ligne produit l'ensemble des noms de genre à plusieurs mots qui ont une correspondance dans le champ species (487 lignes pour 46 valeurs). Une vérification visuelle nous indique des noms latins correctes et "Emplacement libre" (247 lignes très localisées spatialement) qui sort du lot.
Erreurs avec ";"
tableau_selection("\\;")
Ce sont vraisemblablement des erreurs d'encodage. Il n'y a que 4 lignes pour 4 valeurs. Il est difficile de savoir si l'erreur est dans le premier terme ou dans le second et le champs species ne permet pas de trancher (NA).
Voir aussi
Pages internes en lien
- Tag:natural=tree : Traçage d'un arbre seul, souvent isolé ou digne d'intérêt.
- Key:species : Nom scientifique d'une espèce vivante ou fossile.
- Key:denotation : La dénotation est une description utilisée pour indiquer en quoi cette végétation peut être significative.